Ergebnisse
Die enormen Größen und die Zeitabhängigkeit der Daten in der Partikelvisualisierung führen dazu, dass der Datentransfer zwischen Sekundärspeicher, Hauptspeicher und Graphikkartenspeicher untersucht und optimiert werden muss. Mithilfe automatisierter Messungen wurde die Leistungsfähigkeit unterschiedlicher Transfermethoden auf unterschiedlichen Hardwarekonfigurationen ermittelt, um eine optimale Implementierung zu erreichen (Publikation: pvis09-grottel).
|
|
|
Basierend auf den Datensätzen des Teilprojekts B.5 wurde ein zweistufiges Culling-Verfahren entwickelt, welches für Partikeldaten optimiert wurde und auf normalen Desktopcomputern eine interaktive Darstellung von Datensätzen in den Größenordnungen 107 bis 109 erlaubt (Publikation: eurovis10-grottel). Beispielsweise können die Daten einer Simulation einer Laserablation mit 48 Millionen Atomen (Abbildung 2) interaktiv mit etwa 10 FPS dargestellt werden. Die Möglichkeiten, die Darstellungsgeschwindigkeit auf normalen Desktop-Systemen zu optimieren, sind damit weitgehend ausgeschöpft. Zur weiteren Steigerung der Leistung, um beispielsweise noch größere Datensätze interaktiv Visualisieren zu können, soll nun GPU-Cluster-Hardware eingesetzt werden. Hierbei soll unter anderem untersucht werden, inwieweit der nach wie vor bestehende Engpass des Datentransfers mit Hilfe von Lastverteilung auf GPU-Clustern gelöst werden kann.
Um parallel zur Geschwindigkeit auch die Darstellungsqualität zu steigern, wurde die Methode des „Deferred Shading“ erweitert. So können von Atomen gebildete Flächenstrukturen auch dann noch klar und ohne Aliasing dargestellt werden, wenn sich die einzelnen Partikel im Sub-Pixel-Bereich bewegen.
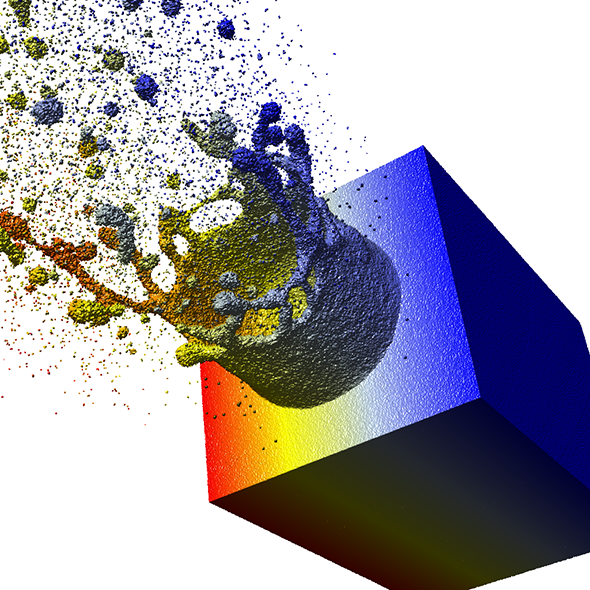
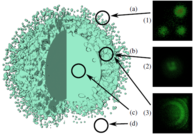
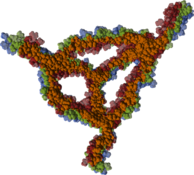
Weiterhin wurde auch untersucht, wie sich die Rendering-Methoden für komplexere Glyphen optimieren lassen. Vorarbeiten zum Dipol-Glyphen (Abbildung 1) dienten als Ausgangspunkt für zusammengesetzte Glyphen von Molekülen ohne innere Freiheitsgrade (Abbildung 3). Diese Moleküldarstellungen können sowohl auf der CPU als auch direkt auf der Graphikkarte erzeugt und so für unterschiedliche Daten optimiert werden. In enger Kooperation mit dem Teilprojekt B.3 wurde auf den bestehenden Erfahrungen aufbauend eine Rendering-Methode für polyederförmige Glyphen geschaffen (Abbildung 4; Publikation: sigrad10-grottel). Hierdurch ist eine von Sampling-Auflösungen unabhängige Visualisierung für poröse Medien geschaffen worden, welche auch für das Teilprojekt A.6 nützlich werden kann. Basierend auf dieser Darstellung werden semi-automatische visuelle Analysemethoden entwickelt.
|
|
Um allerdings die Wahrnehmbarkeit und die Verständlichkeit der Daten zu steigern ist eine visuelle Filterung der Daten sinnvoll und notwendig. Darstellungen mit Kugelglyphen zeigen die Partikeldaten direkt und unverfälscht, als Analyse-Basis und Referenz für Optimierungen. Zur visuellen Analyse der Daten, vor allem der in den Daten enthaltenen Strukturen, ist jedoch eine Extraktion und abstraktere Darstellung dieser Strukturen sinnvoll. Beispielsweise wurde in Kooperation mit Teilprojekt B.2 eine Visualisierung von Fehlstrukturen in Kristallgittern von Metallen durchgeführt (Abbildung 5; Publikation: topoinvis09-grottel). Auf dieser Basis sollen nun weitere Zusammenarbeiten mit den Teilprojekten B.1 und B.7 erfolgen.
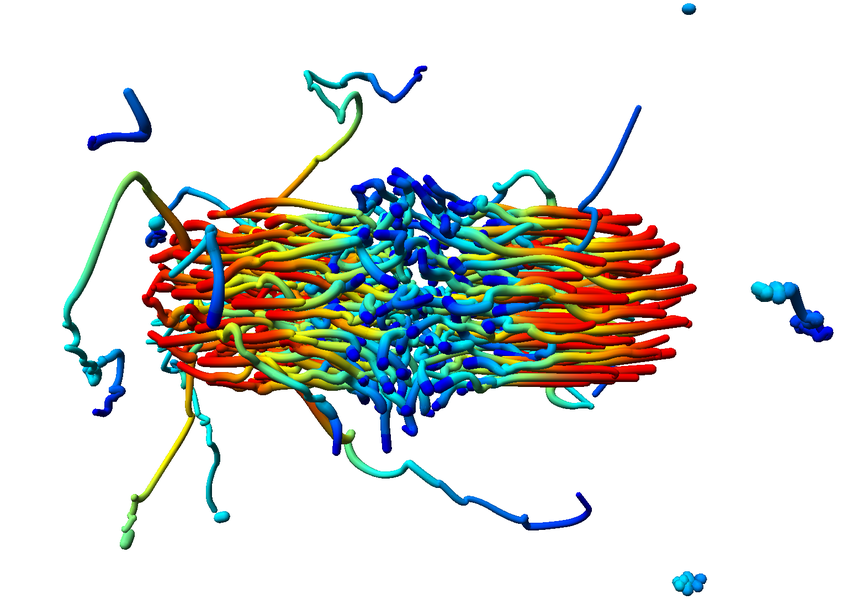
Solche extrahierten Strukturen sind vor allem interessant, wenn die Dynamik und zeitliche Entwicklung innerhalb von Datensätzen untersucht werden soll. Die unverarbeiteten Atomdaten bieten hierbei kaum Möglichkeiten, wohingegen die Entwicklung von abstrakteren Strukturen über der Zeit viele Rückschlüsse, auch auf die verwendeten Algorithmen in Simulation und Analyse, zulassen. In Kooperation mit Teilprojekt A.1 wurde die zeitliche Entwicklung von Molekülcluster in einer Nukleationssimulation mittels visueller Analyse untersucht. Insbesondere die Interaktion mehrerer Molekülcluster untereinander war hierbei ein Schwerpunkt. Damit konnten beispielsweise Probleme mit der Klassifikation dieser Cluster erkannt werden (Publikation: vis07grottel). Ausgehend von dieser Arbeit sollen die zeitlichen Vorgänge in den Datensätzen der Teilprojekte A.8 und B.6 mit ähnlichen Diagrammen über der Zeit untersucht werden.
|
|
|
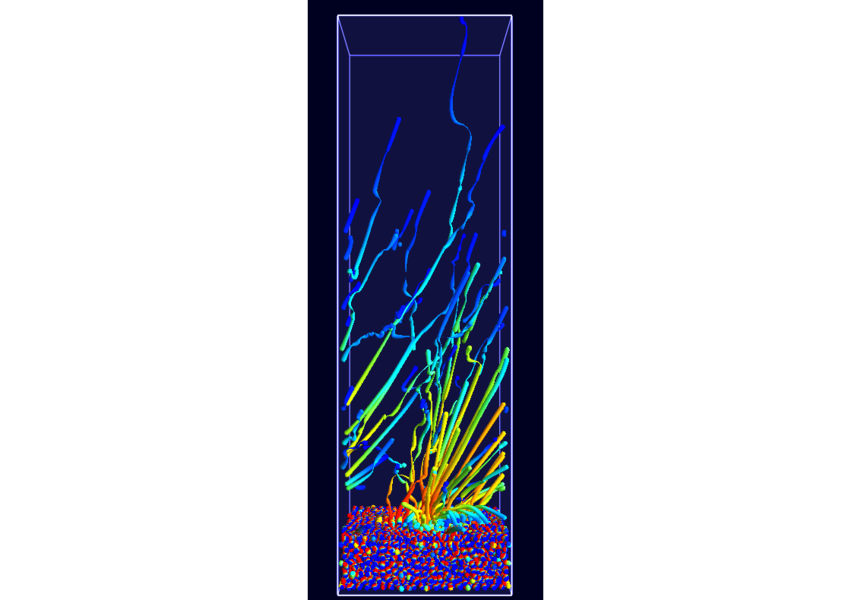
Kombination aus diesen sehr abstrakten Darstellungen von Effekten über der Zeit und räumlich konkreten Darstellungen, wie beispielsweise normalen Pfadlinien, wurde in Kooperation mit A.1 und B.5 eine Möglichkeit entwickelt, die Dynamik eines Datensatzes in statischen Bildern darzustellen. Um die Probleme der Verdeckung und der visuellen Überladung, wie sie bei klassischen Pfadliniendarstellungen auftreten, zu reduzieren, wurde die Menge der darzustellenden Daten zunächst mittels neuer, dichteerhaltender Clusteringverfahren reduziert. Die so entstandene kleinere Menge von Cluster-Partikeln konnte dann über die Zeit hinweg im normalen, physikalischen Raum nachverfolgt werden (Abb. 7; Publikation: pacificvis11-frey). Auf dieser Basis sollen weitere statische Darstellungen von dynamischen Entwicklungen für Daten anderer Teilprojekte entstehen, welche eine deutlich effektivere Analyse ermöglichen sollen.
Durch konsequente Fortführung der Arbeiten konnten auch in der zweiten Förderperiode Fortschritte erzielt werden, sowohl was den Umgang mit großen Datenmengen oder Ensembles als auch eine verbesserte Darstellungsqualität im Allgemeinen betrifft, wodurch die visuelle Analyse komplexer dreidimensionaler Strukturen deutlich erleichtert wurde.
Neue Abstraktionen komplexer Sachverhalte, wie Dipolmomente in Metalloxiden und Wasserstoffbrücken in der Polymer-Lösungsmittelinteraktion wurden erfolgreich publiziert.
Zur Verbesserung der Tiefenwahrnehmung detaillierter Simulationsdatensätze wurde eine hochauflösende Stereoprojektion konzipiert und in den SFB-Projekten zum Einsatz gebracht. In diesem Kontext wurde auch ein generisches Verfahren zur verteilten Berechnung und Anzeige auf diesem Ausgabegerät publiziert.
Als Beitrag zur Grundlagenforschung im Hochleistungsrechnen wurde eine Methode zur Verbesserung der Auslastung von GPUs vorgestellt.
Ein Problem heutiger Grafikprozessoren ist die Divergenz bei Verzweigungen (z.B. if), wenn viele der parallel und synchron arbeitenden Prozessoren unterschiedlichen Code ausführen müssten. Normalerweise wird dieses Problem durch Ausführen aller Alternativen und anschließender Maskierung der entsprechenden Ergebnisse gelöst. Um diese doppelte Ausführung zu vermeiden, wurde ein Verfahren entwickelt, das die Verzweigung von Code auf der GPU durch eine Umgruppierung der Aufgaben in einen Beschleunigungszuwachs umwandelt. Die Aufgaben werden zur Laufzeit ihren Verzweigungen entsprechend gruppiert ausgeführt, um bis auf einen gewissen Verschnitt auf Grund der Warp-Granularität Divergenz komplett zu vermeiden.
Das als anwendungsorientierte Visualisierungsinfrastruktur konzipierte MegaMol™ wird inzwischen auch bei Kollaborationspartnern an den Universitäten von Paderborn, Kaiserslautern und Dresden eingesetzt und eignet sich für die partikelbasierte Visualisierungsforschung im SFB besser als andere frei verfügbare Visualisierungssysteme.
Als neuestes Ergebnis wurde ein graphischer Editor für die dynamische Modulvernetzung und Konfiguration erarbeitet, der es Nutzern erleichtert, die relevanten Module für eine visuelle Analyse zu identifizieren und nur in funktionstüchtiger Weise zu koppeln.
|
|

|
Das Teilprojekt D.3 setzt damit die äußerst erfolgreiche Arbeit aus den ersten beiden Förderperioden fort und strebt weiterhin die ständige Verbesserung bestehender Visualisierungen und die Neuentwicklung von Methoden zur Visualisierung großer, zeitabhängiger Partikeldatensätze an. Die Nützlichkeit der entstehenden Methoden wird auch weiterhin durch enge Kooperationen mit anderen Teilprojekten gewährleistet.
Die Arbeiten der zweiten Förderperiode mündeten im Abschluss der Dissertation des Projektbearbeiters S. Grottel, welche durch den Doktorandenpreis des SFB 716 ausgezeichnet wurde.
Ergebnisse der dritten Förderperiode
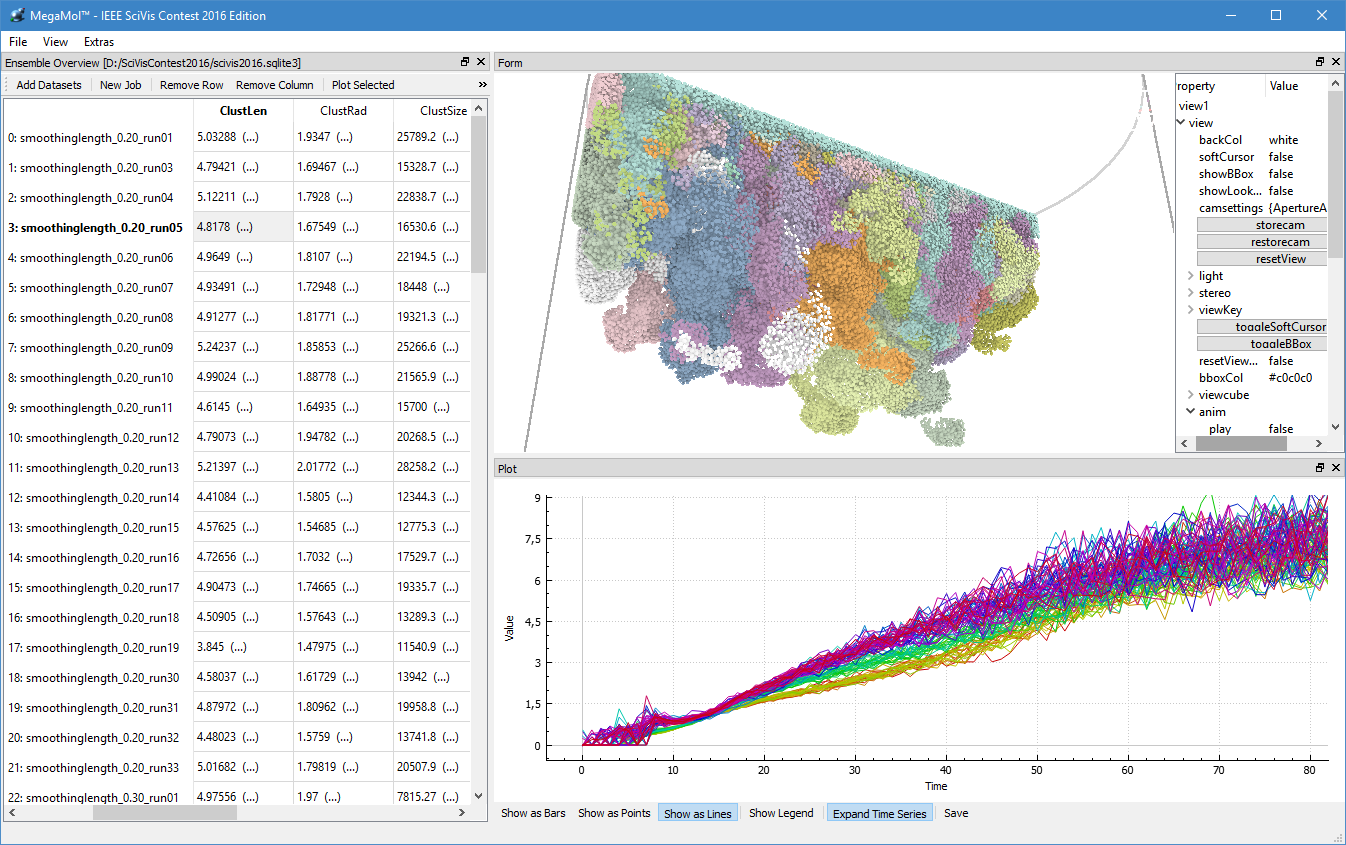
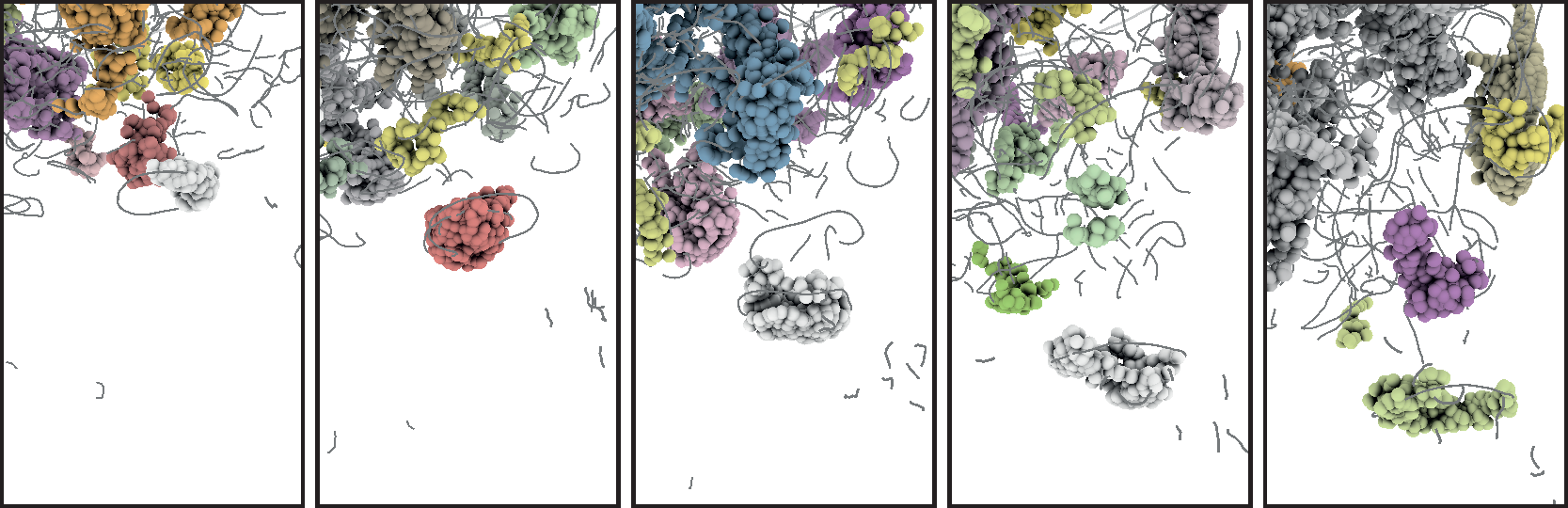
Während der dritten Förderperiode konnten wir gemeinsam mit den Teilprojekten D.3, D.4 und A.6 des SFB 716 sowie weiteren Partnern des CRC 75 und der TU Dresden den IEEE SciVIS Contest 2016 für uns entscheiden. In diesem Wettbewerb bestand die Aufgabe darin, die Eigenschaften von neuentstehenden zähflüssigen Salzkristallen, die sich in Wasser auflösen, in unterschiedlichen Simulationsdurchläufen zu vergleichen. Die kumulierten Eigenschaften wurden in einer Datenbank gesammelt, die der Benutzer über unser Visual Analyse-Programm durchsuchen kann (vlg. Abb. 1 und 2).
|
|
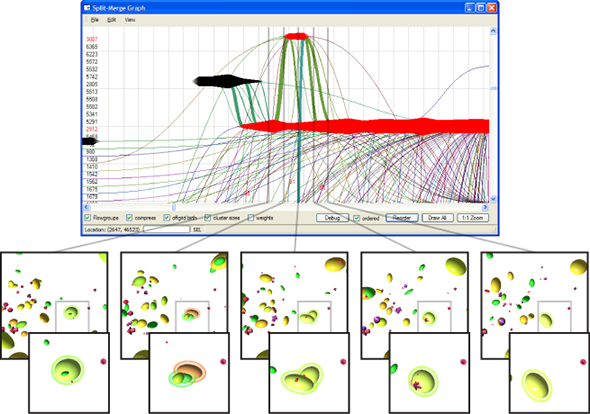
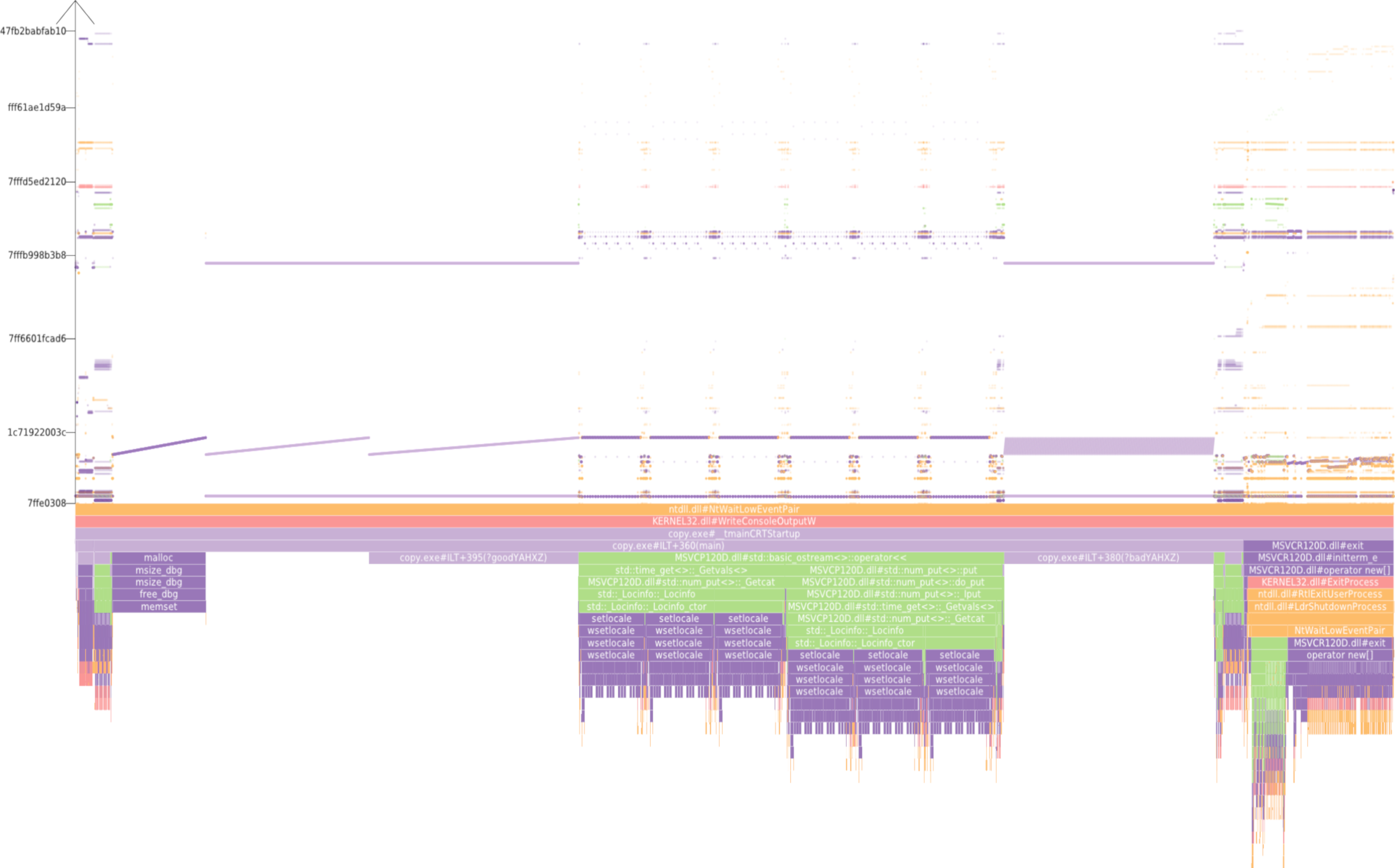
Der Umgang mit großen partikelbasierten Datensätzen macht Datenstrukturen und Algorithmen erforderlich, die den Zugang und die Speicherung der Daten so effizient wie möglich machen – nicht nur in Bezug auf das Rendering und die Möglichkeit der visuellen Analyse. Effizienz zu messen, kristallisiert sich jedoch als schwierige Aufgabe heraus und die Interpretation von statistischen Messungen erschwert es ohnehin, den Code zu optimieren. Um die Effizienz des Speichers der Software, die wir in diesem Teilprojekt verwenden, berechnen zu können, haben wir ein Visual Analyse-Tool (Abb. 2) entwickelt, das Informationen bei jedem einzelnen Speicherzugriff beim Aufrufen einer Applikation erfasst. Diese Zugriffe werden so dargestellt, dass die dahinter liegenden Muster hervorgehoben werden. Sachkundigen Nutzern ist es dadurch unmittelbar möglich, basierend auf den Mustertypen, nachteilige Muster herauszufiltern, um eine höhere Performance zu erreichen. Diese Arbeit wurde mit dem Best Paper auf der VISSOFT 2017 ausgezeichnet.
|